ESPECIES CRÍPTICAS EN Ochlerotatus Taeniorhynchus MEDIANTE REVELACIÓN DE CÓDIGO DE BARRAS
CRYPTIC SPECIES IN Ochlerotatus Taeniorhynchus BY BARCODE DISCLOSURE
Rahuel Jeremías Chan-Chable1, Arely Martínez-Arce2, Pedro Christian Mis Avila1
1 Servicios Estatales de Salud de Quintana Roo. México.
2 El Colegio de la Frontera Sur, Unidad Chetumal. México
Correspondencia: Arely Martínez Arce. Correo electrónico: Esta dirección de correo electrónico está protegida contra spambots. Usted necesita tener Javascript activado para poder verla.
Recibido: 15 de marzo de 2018.
Aceptado: 12 de abril de 2018.
RESUMEN
Introducción. Ochlerotatus taeniorhynchus es un mosquito que habita en zonas costeras y pantanosas de agua salobre. Se distribuye ampliamente en el continente americano. Esta amplia distribución puede estar interrumpida por factores geográficos y ambientales que pueden propiciar su especiación.
Objetivo. Comparar especímenes de Oc. taeniorhynchus de México y Estados Unidos de América (USA) a nivel molecular (DNA barcoding) para mostrar evidencia de la existencia de especies crípticas en esta especie.
Materiales y método. Los especímenes se colectaron con trampas de luz CDC cebadas con dióxido de carbono (CO2). El trabajo de laboratorio molecular se llevó a cabo con protocolos estandarizados. Se usó el programa MEGA para el cálculo de distancias genéticas y el método Automatic Barcode Gap Discovery (ABGD) para delimitar las unidades taxonómicas operativas moleculares (MOTU´s).
Resultados. El análisis de distancias genéticas Kimura 2 parámetros y el árbol Neighbor-Joining mostraron dos clados bien diferenciados de Oc. taeniorhynchus con un valor de divergencia promedio entre clados mayor a 2% (3,1%). Por un lado se agruparon las secuencias de los especímenes provenientes de México y por otro las secuencias de especímenes de USA. Lo anterior también fue soportado por el análisis ABGD.
Conclusión. El valor de divergencia genética promedio de 3,1% observado entre los especímenes de Oc. taeniorhynchus de Mexico y USA indica que se trata de especies crípticas. Se recomienda añadir datos ecológicos y hacer una comparación fina de la morfología de las larvas, el exocorion de huevecillos y genitalia de machos adultos para encontrar las diferencias que las separa como especies.
Palabras clave: Código de Barras del ADN Taxonómico; Complejo IV de Transporte de Electrones; México; Mosquitos Vectores
ABSTRACT
Introduction. Ochlerotatus taeniorhynchus is a mosquito that inhabits coastal and brackish water swamps. It is widely distributed in the Americas. This wide distribution may be interrupted by geographical and environmental factors that may lead to its speciation.
Objective. To compare specimens of Oc. taeniorhynchus from Mexico and the United States of America (USA) at the molecular level (DNA barcoding) to show evidence of the existence of cryptic species in this species.
Materials and method. The specimens were collected with CDC light traps baited with carbon dioxide (CO2). Molecular laboratory work was carried out using standardized protocols. The MEGA program for the calculation of genetic distances and the Automatic Barcode Gap Discovery (ABGD) method were used to delimit the molecular operational taxonomic units (MOTU´s).
Results. The Kimura 2 parameter genetic distance analysis and the Neighbor-Joining tree showed two well-differentiated clades of Oc. taeniorhynchus with an average divergence value between clades greater than 2% (3.1%). On the one hand, the sequences of the specimens from Mexico were grouped and on the other hand, the sequences of specimens from the USA. This was also supported by the ABGD analysis.
Conclusion. The average genetic divergence value of 3.1% observed between the specimens of Oc. taeniorhynchus from Mexico and USA indicates that they are cryptic species. It is recommended to add ecological data and make a fine comparison of the morphology of the larvae, the exocorion of eggs and the genitalia of adult males to find the differences that separate them as species.
Key words: DNA Barcoding, Taxonomic; Electron Transport Complex IV; Mexico; Mosquito Vectors.
INTRODUCCIÓN
Ochlerotatus taeniorhynchus (Wiedemann) es un mosquito importante en medicina humana y veterinaria, es un vector eficiente en la transmisión del virus de la encefalitis equina venezolana (VEFE) tipo epidémico-epizoótico(1,2). En los Estados Unidos ha sido incriminado como vector del virus de la encefalitis equina del Este (EEE) (3) y como vector potencial de la encefalitis del Nilo Occidental (4). Este mosquito ha sido incriminado como el vector primario de la dirofilariasis canina causada por el nematodo parásito Dirofilaria immitis (5).
Las hembras adultas de Oc. taeniorhynchus son muy similares morfológicamente a las de Coquillettidia nigricans (Coquillett) y Cq. venezuelensis (Theobald) por lo que personal con poca experiencia las puede confundir a simple vista. Las tres especies poseen tarsos con bandas de escamas blancas y un anillo de escamas blancas en la probóscide.
Oc. taeniorhynchus es una especie de zona costera, adaptada a hábitats de aguas salobres, abundante y voraz, de hábitos antropófagos diurnos y nocturnos (5,6,7). Esta especie se ha documentado con una amplia distribución en el continente americano, desde Massachusetts en los Estados Unidos hasta Santa Catarina en el sur de Brasil, en la costa atlántica desde California en los Estados Unidos hasta Perú en la costa del Pacífico (1,6,8). También se ha documentado en las costas tanto del Atlántico como del Pacífico de Colombia, en las Antillas y las Islas Galápagos (6,9). Es probable que esta amplia distribución sea interrumpida por barreras físicas y biológicas que pudieran estar generando especiación críptica en ésta especie.
La investigación sobre especies crípticas ha aumentado exponencialmente en las últimas dos décadas, alimentada en gran parte por la creciente disponibilidad y uso de secuencias de ADN (10,11,12). Se han documentado la presencia de especies crípticas o complejos de especies en culícidos de los géneros Aedes, Anopheles y Culex (13,14).
La identificación correcta de las especies de culícidos, así como el descubrimiento de especies crípticas es esencial para la elaboración de programas de control y prevención de las enfermedades que transmiten las especies vectores (10,15,16).
El objetivo de este estudio fue analizar secuencias del gen Citocromo Oxidasa subunidad I (COI) generadas en este estudio y aquellas generadas en el 2012 por Gibson y colaboradores (17) para mostrar evidencia de especies crípticas en Oc. taeniorhynchus. Además construir un árbol de identidad basado en las distancias genéticas para conocer si los especímenes de ambos países se agrupan en un mismo clado.
MATERIALES Y MÉTODO
Colecta e identificación de mosquitos
En octubre de 2013, fueron colectadas hembras adultas de Oc. taeniorhynchus con el uso de trampas de luz tipo CDC (Centers for Disease Control and Prevention) operando consecutivamente en la ciudad de Chetumal, Quintana Roo, Mexico de 18:00 hrs a 24:00 hrs. Cuatro sitios fueron seleccionados: 1) Locación La Sabana, 18°31´7.5´´N–88°21´6.04´´W; 2) Locación Parque Ecológico, 18°30´25.4´´N–88°19´13.0´´W; 3) Locación Bahía, 18°29´46.03´´N–88°18´55.26´´W; y 4) Locación El Colegio de la Frontera Sur (ECOSUR), 18°32´35.0´´N–88°15´43.9´´W (figura 1). Se usaron 2 trampas por sitio, las cuales fueron cebadas con CO2 y colgadas a 40 cm sobre el nivel del suelo. Todos los especímenes colectados fueron depositados en la colección entomológica del museo de Zoología de ECOSUR, unidad Chetumal, código de referencia ECO-CH-AR/DP_0096-0155.
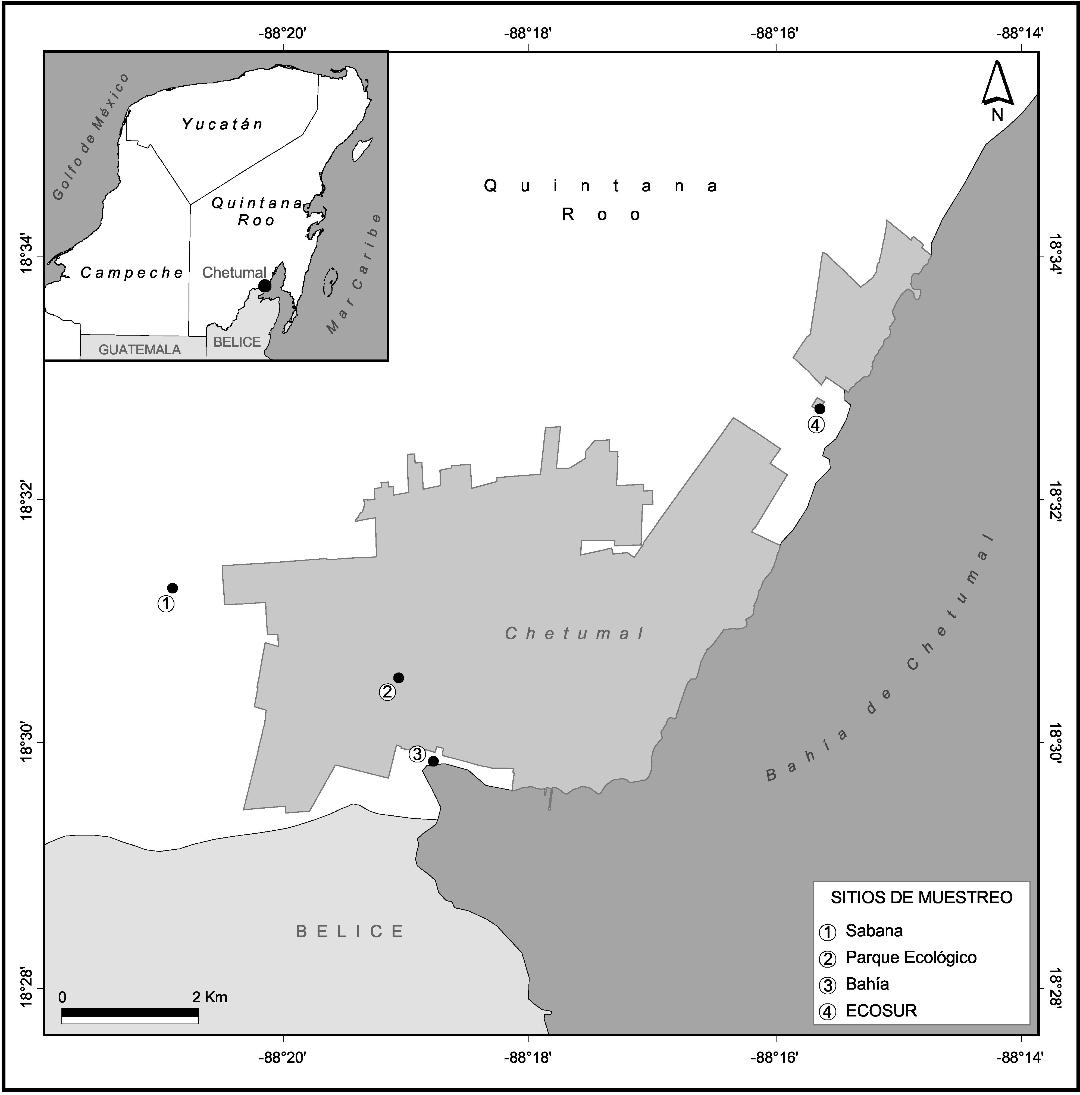
Figura 1. Localidad y sitios de colecta de Oc. taeniorhynchus en Quintana Roo.
Todos los ejemplares fueron montados y fotografiados bajo un estereoscopio Zeiss Germany Stemi SV 6 con una cámara Nikon Sigma 50mm F2.8 EX DG Macro. Para la identificación morfológica de las hembras adultas se usaron las claves de identificación de los mosquitos de Guatemala (18). Los datos de colecta están despositados en el proyecto ‘‘Culicids of Chetumal Mexico’’ (CUL) en BOLD Systems® (19).
Análisis molecular
Para el análisis molecular (Código de barras de ADN), se usó el gen citocromo oxidasa subunidad I (COI). Diez ejemplares de Oc. taeniorhynchus fueron seleccionados para ser procesados. El ADN fue extraído de acuerdo a Ivanova et al. 2006 (20), la amplificación se realizó con el mix de PCR en un volumen final de 12,5 µl, conteniendo 2 µl de ADN templado y usando los primers LCO1490/HCO2198 (21, 22). Las secuencias fueron editadas en el programa Codon Code Aligner v 5.0.1.
Para proporcionar una representación gráfica del patrón de agrupamiento entre las secuencias de Oc. taeniorhynchus de Mexico y USA el software MEGA v. 6.06 (23) fue usado y fue construido un árbol de similitud Neighbor-Joining (NJ) basado en distancias genéticas usando el modelo de distancia Kimura 2 parámetros (K2P) (24, 25). Además fueron calculadas las distancias intraespecíficas e interespecíficas (p) con MEGA v. 6.06 (23).
El árbol de Oc. taeniorhynchus fue construido con secuencias de México generados en este estudio y USA disponibles en las bases de datos moleculares (Genbank y Bold Systems). Para darle mayor soporte al análisis se usaron secuencias de las especies Oc. Angustivittatus, Oc. fulvus (n = 2) y Oc. serratus (n = 2) dichas especies pertenecen al mismo género al que pertenece Oc. taeniorhynchus por lo que su inclusión ayudara a la separación de los grupos, para cada especie se incluyó dos secuencias (n=6) las cuales fueron generadas en este estudio. Como grupo externo se utilizó la secuencia de Anopheles albimanus.
Para delimitar las unidades taxonómicas operativas moleculares (MOTU's) se utilizó el programa ABGD (Automatic Barcode Gap Discovery) (26), con una distancia intraespecífica mínima (Pmin) de 0.001, una distancia intraespecífica máxima (Pmax) que oscila de 0.02 a 0.1 y el parámetro barcode gap width con la configuración predeterminada (1.5) y los modelos evolutivos K2P y Jukes-Cantor (JC) (27). El análisis fue realizado para las secuencias de Oc. taeniorhynchus obtenidas en este estudio y aquellas descargadas de las bases de datos.
RESULTADOS
Se colectaron un total de 60 hembras adultas de Oc. taeniorhynchus (figura 2). El Colegio de la Frontera Sur (ECOSUR) con 29 especímenes fue el sitio que presentó la mayor abundancia de Oc. taeniorhynchus, mientras que la Bahía con 2 ejemplares fue la que presentó la menor abundancia. De los 10 ejemplares de Oc. taeniorhynchus procesados para este estudio se logró generar 6 secuencias del gen COI para el estado de Quintana Roo, México. Las secuencias genómicas obtenidas están depositadas en la base de datos de BOLD Systems® (19) dentro del proyecto ‘‘Culicids of Chetumal Mexico’’ (CUL).

Figura 2. Hembra adulta de Oc. taeniorhynchus: (A) vista lateral, (B) vista dorsal.
En cuanto a la búsqueda en las bases de datos moleculares, 13 secuencias fueron recuperadas, 3 para Mexico y 10 para USA (ver cuadro 1). Los análisis moleculares mostraron que las seis secuencias generadas en Quintana Roo y las secuencias de especímenes de México provenientes de otros Estados corresponden a una sola especie (Oc. taeniorhynchus) con un valor de divergencia intraespecífica promedio de 0.2%; y las secuencias de USA corresponden a una segunda especie (ver figura 3). La distancia genética K2P interespecífica promedio de la comparación de las diferentes especies del mismo género (Ochlerotatus) fue > 2% (8.58 %).
Cuadro 1. Secuencias de Oc. taeniorhynchus descargadas de BOLD Systems y GenBank que fueron agregados al análisis en este estudio.
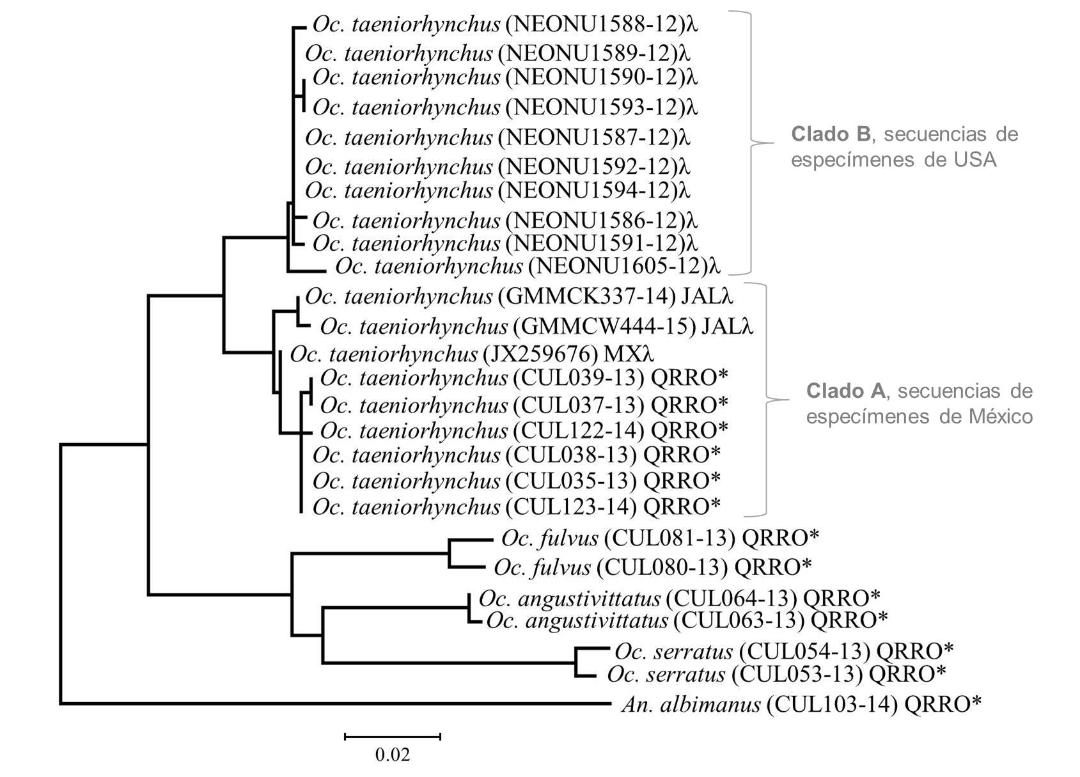
Figura 3. Árbol Neighbor-joining basado en las distancias genéticas (K2P) del gen COI. Las secuencias generadas en este estudio se señalan con un asterisco (*). Las secuencias de GenBank se señalan por el símbolo lambda (λ), y las de Bold systems por el símbolo yen (¥). Las abreviaciones JAL = Jalisco, QRRO = Quintana Roo, y MX = México.
El árbol NJ de distancias genéticas mostró dos clados bien diferenciados de Oc. taeniorhynchus con un valor de distancia promedio entre clados > 2% (3,1%) y una distancia máxima observada de 3,5%. Las secuencias de los especímenes provenientes de Quintana Roo y las secuencias pertenecientes a Jalisco forman el clado A y las secuencias de USA del estado de Florida forman el clado B (figura 3).
El método ABGD identificó 2 MOTU's para las 19 secuencias de Oc. taeniorhynchus (cuadro 3). Las secuencias de especímenes de Quintana Roo y de Jalisco fueron agrupados en un mismo MOTU y las secuencias de Florida USA fueron agrupadas en otro MOTU.
Cuadro 3. MOTU´s recuperados de COI de Oc. taeniorhynchus por el método ABGD en este estudio. * = divergencia intraespecífica a priori (P); I = particiones iniciales; R = particiones recursivas. El número de particiones ABGD obtenidas por los modelos JC y K2P fue el mismo. El parámetro Relative Gap Width (X) fue 1.5.
DISCUSIÓN
Ae. taeniorhynchus es una especie dominante que presenta una amplia plasticidad ecológica pudiendo ser encontrada en zonas urbanas, rurales y selváticas (28). Su alta abundancia en ECOSUR puede ser debida a las condiciones ambientales de humedad y temperatura favorables presentes en el sitio. Hasta el momento en el estado de Quintana Roo solo se ha reportado como molestia sanitaria sobretodo en la temporada de lluvias cuando su abundancia presenta el pico más elevado en los meses de septiembre y octubre.
Por otro lado, todos los ejemplares analizados han sido identificados morfológicamente como Oc. Taeniorhynchus; sin embargo, el valor promedio de divergencia genética de 3,1% observada muestra claramente la separación de 2 clados sugiriendo la presencia de especies crípticas dentro de Oc. taeniorhynchus.
Un estudio previo realizado por Gibson et al. 2012 (17), con ejemplares de Oc. taeniorhynchus de USA y un ejemplar de México, menciona una divergencia mayor al 2% (2,18%) entre los ejemplares analizados. Sin embargo, ellos no pudieron corroborar que se trataba de especies distintas, ya que solo contaban con una secuencia de un espécimen de México el cual se separaba de los ejemplares de USA.
La presencia de especies crípticas o complejos de especies ha sido señalado en otros géneros de culícidos como Aedes, Anopheles y Culex (13,14). Wang et al. (2012) (13) observaron una distancia K2P máxima de 4,71% en ejemplares de Cx. modestus colectados en China. Ellos no encontraron variaciones en la morfología de las hembras adultas pero sí en las setas del tórax de las larvas y en la genitalia de los machos adultos. Así mismo Wang et al. (2012) (13), también observaron una distancia intraespecífica > 2,0% (2,61%) en An. sinensis y aunque ellos no observaron diferencias morfológicas dentro de esta especie, las diferencias en los hábitos de alimentación y hábitat han sido ampliamente documentados (29).
En el presente estudio se han analizado un mayor número de secuencias que evidencian la presencia de más de una especie de Oc. taeniorhynchus, una especie para Mexico y una especie diferente para USA. Lo anterior también es apoyado por el análisis ABGD en el que se observaron dos MOTU’s, una que agrupó a las secuencias de ejemplares de México y otra que agrupo a las secuencias de especímenes de USA.
Son necesarios futuros análisis que involucren un muestreo a lo largo de México y del continente americano, pues debido a la amplia distribución de Oc. taeniorhynchus se podría considerar la existencia de más especies crípticas. Es necesario analizar a detalle los especímenes de México y USA, tomando en cuenta la morfología de las etapas inmaduras (huevos, larvas y pupas), la morfología de la genitalia de los machos adultos, los factores físicos (barreras geográficas, humedad y temperatura) y datos biológicos (30, 31) para encontrar el o los caracteres que los separa como especies y así poder hacer la descripción de la nueva especie.
CONCLUSIONES
Los códigos de barra de ADN mostraron evidencia de la existencia de especies crípticas en Oc. taeniorhynchus. Un clado es formado por las secuencias COI de especímenes pertenecientes a diferentes estados de México, y otro clado formado por secuencias de especímenes de USA. Además de la información biogeográfica y los datos moleculares, se recomienda añadir datos ecológicos y realizar la comparación de la morfología de larvas, el exocorion de huevecillos y genitalia de machos adultos para encontrar las diferencias que las separa como especies.
Los códigos de barra de ADN son una herramienta que permiten descubrir especies crípticas y determinar el máximo nivel de identidad la mayoría de las especies, por lo que se considera una herramienta complementaria a la taxonomía tradicional.
Agradecimientos
Agradecemos a Francisco R. Castañeda-Rivero por su apoyo en las salidas de muestreo, a Humberto Bahena-Basave por la elaboración de imágenes, a Holger Weissenberger por la construcción del mapa de los sitios de colecta, y a Noemí Salas Suárez por los materiales de laboratorio entomológico. También agradecemos a la Red MEXBOL proyecto no. 271108 financiado por el Consejo Nacional de Ciencia y Tecnología.
REFERENCIAS
- Forattini OP. Entomología Médica. 1ª ed. Sao Paulo, Brasil: Editora da Universidad da Sao Paulo; 1965.
- Brault AC, Powers AM, Ortiz D, Estrada-Franco JG, Navarro-Lopez R, Weaver SC. Venezuelna equine encephalitis emergence: Enhance vector infection from a single amino acid substitution in the envelope glycoprotein. Proc. Natl. Acad. Sci. USA 2004; 101:11344-11349.
- Ortiz DI, Wozniak A, Tolson MW, Turner PE, Vaughan DR. Isolation of EEE virus from Ochlerotatus taeniorhynchus and Culiseta melanura in coastal South Carolina. J. Am. Mosq. Control Assoc. 2003; 19: 33-38.
- Hribar LJ, Vlach JJ, Demay DJ, Stark LM, Stoner RL, Godsey MS, et al. Mosquitoes infected with West Nile virus in the Florida Keys, Monroe County, Florida, USA. J. Med. Entomol. 2003; 40: 361-363.
- Manrique-Saide P, Escobedo-Ortegón J, Bolio-González M, Sauri-Arceo C, Dzib-Florez S, Guillermo-may G, et al. Incrimination of the mosquito, Aedes taeniorhynchus, as the primary vector of heartworm, Dirofilaria immitis, in coastal Yucatan, Mexico. Med. Vet. Entomol. 2010; doi: 10.1111/j.1365-2915.2010.00884.x
- Olano VA. Hallazgo de Aedes taeniorhynchus (Wiedemann 1821) en un lugar del municipio de Ambalema, Departamento del Tolima (Colombia) (Diptera: Culicidae). Biomédica 1985; 5: 26-28.
- Mankin RW. Acoustical detection of Aedes taeniorhynchus swarms and emergence exoduses in remote salt marshes. J. Am. Mosq. Control Assoc. 1994. 10: 302-308.
- Walter Reed Biosystematics Unit. [internet]. Washington, DC: Smithsonian Institution [acceso 25-04-2018]. WRBU Systematic catalog of Culicidae. Disponible en: http://www.mosquitocatalog.org/main.asp.
- Bello F, Ruiz-García M. Isoenzyme Polymorphism and Genetic Structure of Ochlerotatus taeniorhynchus (Diptera: Culicidae) in Populations from the Colombian Atlantic Coast. Biochem Genet. 2009; 47: 462-470.
- Bickford D, Lohman DJ, Sodhi NS, Ng PKL, Meier R, Winker K, et al. Cryptic species as a window on diversity and conservation. Trends Ecol. Evol. 2007; 22(3): 148-155. https://doi.org/10.1016/j.tree.2006.11.004.
- Sáez AG. Genes y especies. . Ecosistemas 2009; 18: 3-9.
- Kress WJ, García-Robledo C, Uriarte M, Erickson DL. DNA barcodes for ecology, evolution, and conservation. Trends Ecol. Evol. 2014; 30: 1-11.
- Wang G, Li C, Guo X, Xing D, Dong Y, Wang Z, et al. Identifying the Main Mosquito Species in China Based on DNA Barcoding. PLoS ONE 2012; 7. https://doi.org/10.1371/journal.pone.0047051.
- Ashfaq M, Hebert PDN, Mirza JH, Khan AM, Zafar Y, Mirza MS. Analyzing Mosquito (Diptera: Culicidae) Diversity in Pakistan by DNA Barcoding. PLoS ONE 2014; 9(5): e97268. https://doi.org/10.1371/journal.pone.0097268.
- Paredes-Esquivel C, Donnelly MJ, Harbach RE, Townson H. A molecular phylogeny of mosquitoes in the Anopheles barbirostris Subgroup reveals cryptic species: Implications for identification of disease vectors. Mol. Phylogenetics Evol. 2009; 50: 141-151.
- Azari-Hamidian S, Linton YM, Abai MR, Ladonni H, Oshaghi MA, Hanafi-Bojd AA, et al. Mosquito (Diptera: Culicidae) fauna of the Iranian islands in the Persian Gulf. J. Nat. Hist. 2010; 44(15): 913-925.
- Gibson CM, Kao RH, Blevins KK, Travers PD. Integrative Taxonomy for Continental-Scale Terrestrial Insect Observations. PLoS ONE 2012; 7(5): e37528. doi:10.1371/journal.pone.0037528.
- Clark-Gil S, Darsie RF. The mosquitoes of Guatemala, their identification, distribution and bionomics. Mosq Syst 1983; 15:151-284.
- Ratnasingham S, Hebert PDN. BOLD: the barcode of life data system (http://www.barcodinglife.org). Mol Ecol Notes 2007; 7: 355-364.
- Ivanova NV, deWaard J, Hebert PDN. An inexpensive, automation-friendly protocol for recovering highquality DNA. Mol Ecol Notes 2006; 6: 998-1002.
- Folmer O, Black M, Hoeh W, Lutz R, Vrijenhoek R. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates. Mol. Mar. Biol. Biotechnol. 1994; 3: 294-299.
- Chan-Chable RJ, Ortega-Morales AI, Martínez-Arce A. First record of Psorophora albipes in Quintana Roo, México. J. Am. Mosq. Control Assoc. 2016; 32(3): 237-239. 10.2987/16-6580.1.
- Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6: Molecular Evolutionary Genetics Analysis Version 6.0. Mol. Biol. Evol. 2013; 30: 2725-2729.
- Saitou N, Nei M. The neighbour-joining method: a new method for reconstructing phylogenetic trees. Mol. Biol. Evol. 1987; 4: 406-425.
- Hajibabaei M, Janzen DH, Burns JM, Hallwachs W, Hebert PDN. DNA barcodes distinguish species of tropical Lepidoptera. Proc. Natl. Acad. Sci. U.S.A. 2006; 103: 968-971.
- Puillandre N, Lambert A, Brouillet S, Achaz G. ABGD, Automatic Barcode Gap Discovery for primary species delimitation. Mol. Ecol. 2012; 21: 1864-1877. 10.1111/j.1365-294X.2011.05239.x.
- Jukes TH, Cantor CR. Evolution of protein molecules. En Munro HN. (editor). Mammalian Protein Metabolism. 1ª ed. New York, USA: Academic Press; 1969. p. 21-132).
- Diéguez Fernández L, Mentor Sarría V, Peña Rodríguez J, Rivero Camejo M. Presence of Culicidae family in the touristic zone of Sta Lucia, Camagüey and its relationship with diseases of medical-veterinary importance. Rev. AMC 2005; 9(2), 1-11.
- Baolin L. Fauna Sinica, Insecta Vol.9, Diptera: Culicidae I, II. Beijing: Science Press; 1997.
- Hebert PDN, Penton EH, Burns JM, Janzen DH, Hallwachs W. Ten species in one: DNA barcoding reveals cryptic species in the neotropical skipper butterfly Astraptes fulgerator. PNAS 2004; 101: 14812-14817.
- Cywinska A, Hunter FF, Hebert PDN. Identifying Canadian mosquito species through DNA barcodes. Med. Vet. Entomol. 2006; 20: 413-424.




