Conocimiento actual de la fisiopatología, diagnóstico molecular y serológico de COVID-19.
Current understanding of the pathophysiology, molecular and serological testing of COVID-19.
Jorgelina Barrios De Tomasi(1,2), María Daniela Pech Vázquez(3)
1 ProfesoraInvestigadora, División de Ciencias de la Salud, Universidad de Quintana Roo
2 Laboratorio Estatal de Salud Pública, Secretaría de Salud del Estado de Quintana Roo
3 Estudiante de tercer semestre de la Licenciatura de Medicina, División de Ciencias de la Salud, Universidad de Quintana Roo.
Correspondencia: Dra. Jorgelina Barrios De Tomasi. Correo electrónico: Esta dirección de correo electrónico está protegida contra spambots. Usted necesita tener Javascript activado para poder verla.
Recibido: Octubre de 2020
Aceptado: Noviembre de 2020
RESUMEN
A finales del 2019, un nuevo virus emergió en la ciudad de Wuhan, China, el cual se esparció rapidamente convirtiéndose en una pandemia. El agente causante es el coronavirus del sindrome respiratorio severo agudo (SARS-CoV-2), el cual presenta una secuencia genómica diferente a la de otros coronavirus conocidos. El SARS-CoV-2 es un virus con una envoltura con forma de corona y un genoma conformado por una hebra en sentido postivo de ARN. El diagnóstico temprano del COVID-19 ha sido un desafío para los investigadores y trabajadores del área de la salud, ya que las técnicas actuales difieren en su especificidad y sensibilidad dependiendo del estadio de la enfermedad. En este trabajo resumimos las técnicas diagnósticas con sus ventajas y desventajas utilizadas para su diagnóstico.
Palabras clave:COVID-19, SARS-CoV-2, Coronavirus, Fisiopatología del COVID-19.
ABSTRACT
At the end of 2019, a novel coronavirus emerged in Wuhan, China, then spread rapidly throughout the world. The causative agent is severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2), a large virus with different genome from other kind of known coronavirus. Rapid and early diagnosis of COVID-19 is a challenging issue for healthcare personnel. The sensitivity and specificity of the laboratory tests used to diagnose COVID-19 are variable and differ in eficacy depending on the disease’s stage. This work shows the actual understanding of the pathophysiology and diagnosis of COVID-19.
Keywords: COVID-19, SARS-CoV-2, Coronavirus, COVID-19 pathophysiology.
INTRODUCCIÓN
Todos los organismos vivos que existen en nuestro planeta están regidos por los mismos principios biológicos, maquinarias bioquímicas, mensajes químicos y se rigen bajo el mismo código genético. Por lo que aunque pensemos que como seres humanos somos el número uno en la historia evolutiva, no lo somos. Somos igual de susceptibles que cualquier otra especie animal o vegetal que existe en la Tierra. De tal manera que en Biología se utilizan modelos animales para explicar procesos fisiológicos humanos(1).Por ejemplo, la mutación del gen kit (o c-kit) provoca una disminución en la pigmentación de la piel y el cuero cabelludo en la región frontal de la cabeza de una persona. Lo que es interesante es que la mutación de este mismo gen en diversas especies animales tendrá las mismas consecuencias, es decir la despigmentación de la piel en las mismas regiones(1).Así, podemos observar cómo los seres vivos sean animales plantas o humanos, somos semejantes y susceptibles a lo que suceda en nuestro entorno. Esto ha sido claro en estos momentos, donde nos hemos visto afectados por una pandemia ocasionada por un virus. Solamente el 0.1% de pequeñas variaciones en la secuencia de nuestro ADN marcan las diferencias entre los humanos. Alrededor de4% de variaciones marcan la diferencia entre un humano y un chimpancé(2).Un ratón tiene alrededor de 90% de similitud y una banana alrededor de 60%3.Todos los seres vivos compartimos la misma química del agua y de sus macromoléculas. A su vez, las funciones esenciales de la vida son las mismas en los organismos vivos(1,2).
En los años 70s tras el descubrimiento de la codificación de la herencia genética en la doble hélice del ADN se explicaron a su vez, los mecanismos “unidireccionales” de transmisión de la información genética(4). La información de un individuo se encuentra contenida en los genes de una célula. Un gen es una sección de ADN que codifica la información necesaria para crear una proteína. La información genética fluyede los genes del ADN (molécula que almacena todo el genoma de un individuo) donde se realiza una copia en una molécula llamada ARNm mediante un mecanismo llamado transcripción (en el núcleo de la célula). Luego, la información del ARNm se traduce o interpreta con la ayuda de los ribosomas en proteínas (ya sea en el citoplasma o en el Retículo Endoplásmico Rugoso de la célula). Se piensa que todas las proteínas de una célula se producen de esta manera (a excepción de algunas proteinas mitocondriales). Las proteínas son macromoléculas elementales que llevan a cabo los procesos y funciones celulares(4).El ADN a su vez puede duplicarse en un proceso llamado replicación para transmitir la información genética a la descendencia durante la división celular(4). A este flujo de la información unidireccional se le conoce como el dogma central de la Biología Molecular, descrito por Francis Crick. Sin embargo, hoy sabemos que la ciencia es un ente dinámico que no se asienta en dogmas. De este modo, en la era post-genómica han surgido descubrimientos que han permitido re-postular y ampliar este dogma central, demostrando que el flujo de la información puede ir en varios sentidos(5). Inclusive este flujo de la información explica cómo un pequeño virus altera el comportamiento de células sanas y puede ser utilizado para el diagnóstico de la enfremedad.
El objetivo de este trabajo es resumir la basta información que actualmente se ha publicado en relación al coronavirus tipo 2 del sindrome respiratorio agudo grave (SARS-CoV-2); haciendo énfasis en su estructura, fisiopatología y las pruebas que se emplean para su diagnóstico con sus ventajas y desventajas.
¿Qué son los virus?
Los virus son agentes infecciosos microscópicos que requieren de la maquinaria de otro organismo para poder replicarse. A un virus, no se le considera un organismo vivo debido a esta incapacidad de dividirse por sí mismos, aunado a que no tienen un metabolismo y utilizan la energía generada por la célula hospedera. Al infectar una célula, transmiten sus genes y utilizan la maquinaria celular para sintetizar sus ácidos nucleicos y sus proteínas para formar nuevos virus(6).
El SARS CoV-2 es un virus que pertenece a la subfamilia Orthocoronavirinae dentro de la familia de los coronavirus. Existen cuatro géneros en esta subfamilia: Alphacoronavirus, Betacoronavirus,Deltacoronavirusy Gammacoronavirus(7,8).Las dos primeras subfamilias infectan al ser humano, mientras que las dos últimas infectan a aves(8).El SARS CoV-2 se clasifica dentro de los betacoronavirus y es el séptimo virus conocido dentro de este género, que incluye a SARS CoV-1, el cual originó la epidemia del síndrome respiratorio agudo grave, en el 2002 en China e incluye al MERS CoV, que provocó el síndrome respiratorio de Oriente Medio en el 2012 en Arabia Saudita(7,8).Estas tres patologías similares son de origen zoonótico, es decir, que se transmiten de forma natural de los animales a los seres humanos(9).
Fisiopatología del SARS-CoV-2
Para comprender las fisiopatología del SARS-CoV-2 es necesario describir su estructura, su genoma y su ciclo de replicación. El SARS-CoV-2 tiene una estructura esférica de alrededor de 50 a 200 nm en diámetro, la cual esta dada por su envoltura lipídica y proteica (similar a las membranas de las células). Debajo de la envoltura se encuentra una estructura llamada cápside, la cual es una estructura proteica formada por monómeros llamados capsómeros. En el interior de la cápside se encuentra el material genético del virus. El término nucleocápside se utiliza para denotar el material genético de origen viral envuelto en su cápside proteica. El genoma viral en el interior está formado por una cadena de ARN monocatenario de sentido positivo, esto es, que se puede leer en sentido de izquierda a derecha(10,11).Su genoma está constituido por alrededor de 29.9 kb de longitud, un genoma de los más largos conocidos para un virus. Su ARN tiene un capuchón en el extremo 5’ y una cola poli-A en el extremo 3’ (semejante al ARNm de cualquier mamífero) y tiene a su vez 14 marcos abiertos de lectura u ORFs (Open Reading Frames) que codifican para 27 proteinas. Entre estas encontramos las proteínas estructurales que forman parte de la envoltura y la cápside. Las más relevantes se listan a continuación(10,11):
- La proteína S (Spike=púa en inglés). En fotografias de microscopía electrónica, esta proteina forma estructuras parecidas a púas o espinas en su superficie que le dan un aspecto similar a una corona dándole su nombre a los coronavirus. Esta proteína es vital para la unión del virus a la célula hospedera. Es una glicoproteína conformada por una porción extracelular y una intracelular. El dominio extracelular de esta proteína es el que permite la unión al receptor de ACE-2 (enzima-2 convertidora de angiotensina), el dominio intracelular de la proteina contiene la maquinaria necesaria para que el virus se fusione con la membrana de la célula hospedera(11,12).
- La proteína E (Envoltura). Es una pequeña proteína involucrada en el ensamblado, maduración y salida del virus (exocitosis)(10,13).
- La proteína M (Membrana)(10,13).
- La proteína N (Nucleocápside). Es una fosfonucleoproteína que protege al genoma viral cuando esta fuera de su envoltura(10,13).
- La proteina hemaglutinin-esterasa (HE), esta proteína facilita la entrada y la patogénesis del virus(8).
La infección con SARS-CoV-2 es una infección básicamente respiratoria, que se transmite a través de las secreciones respiratorias de las personas infectadas mediante la expulsión de pequeñas gotas de saliva y aerosoles a través del habla, la tos y el estornudo, propiciando así la transmisión del virus (14,15). También se transmite por el contacto directo con estas secreciones en superficies u objetos contaminados o fómites(16).
Cuando estas secreciones contaminadas llegan a la superficie de nuestras mucosas (boca, nariz y ojos), el virus invade hasta llegar a las vías respiratorias bajas(16). Si observamos como es un pulmón en su estructura microscópica, se observan estructuras similares a globos que se llenan de aire durante la aspiración y se vacían durante la espiración. Estos pequeños globos se llaman alveolos y son el principal blanco para SARS-CoV-2. Los alveolos están conformados histológicamente por dos tipos de células epiteliales: las células alveolares (neumocitos) tipo I y las células alveolares (neumocitos) tipo II. Estas células difieren en su estructura y función. Los neumocitos tipo I son las más abundantes, tienen una forma plana cuya función es el intercambio de gases. Por otro lado, los neumocitos tipo II son células con un aspecto esférico y su función es la producción del factor surfactante, una delgada capa de fosfolípidos que recubre el alveolo para evitar el colapso alveolar durante la espiración. Una vez que llega el virus a los alveolos pulmonares inicia su ciclo replicativo(12).
Ciclo replicativo de SARS-CoV-2
El ciclo replicativo de un virus describe el proceso de multiplicación dentro de una célula infectada. La replicación viral del SARS-CoV-2 podemos resumirla en varios pasos(13):
1) Unión del virus a la célula hospedera y entrada
Los neumocitos tipo II tienen en su superficie un receptor transmembranal específico para ACE-2(14). Este receptor en un individuo sano, cumple ciertas funciones como vasodilatador con propiedades antioxidantes y antiinflamatorias, juega a su vez, un papel protector en diversos órganos del sistema cardiovascular, incluyendo al corazón, vasos sanguíneos y riñones(14). Sin embargo, durante una infección viral respiratoria, como SARS-CoV-2, este receptor ACE-2 es la vía de entrada del virus al interior de la célula. El Receptor (ACE-2) se une específicamente a la proteína S del SARS-CoV-2 con una gran afinidad, y es aquí donde se desencadena la infección(12,13). (Fig.1)
Se han descrito dos mecanismos para la entrada del virus (Fig.1)(15). En ambos mecanismos la via de entrada es mediante la unión de la proteína S al receptor ACE-2. El primer mecanismo, una vez que la proteína S del virus se une al receptor ACE-2 de la célula hospedera, la proteína S va a ser cortada por una proteasa transmembranal de tipo serina 2 (TMPRSS2) presente en la membrana de la célula hospedera, quien no identifica al virus como una partícula extraña(12). La envoltura viral entonces, se fusiona con la membrana plasmática liberando así el genoma viral dentro de la célula. En el segundo mecanismo, la proteína S se une al receptor ACE-2 de la célula hospedera y es introducido a la célula por un proceso de endocitosis mediante una pequeña vesícula llamada endosoma. Posteriormente la proteína S, va a ser cortada por una proteasa endosomal llamada catepsina L, lo cual inicia la fusión de la envoltura viral con la bicapa lipídica del endosoma, liberando el genoma viral al interior de la célula(12). (Fig.1)
Figura 1.
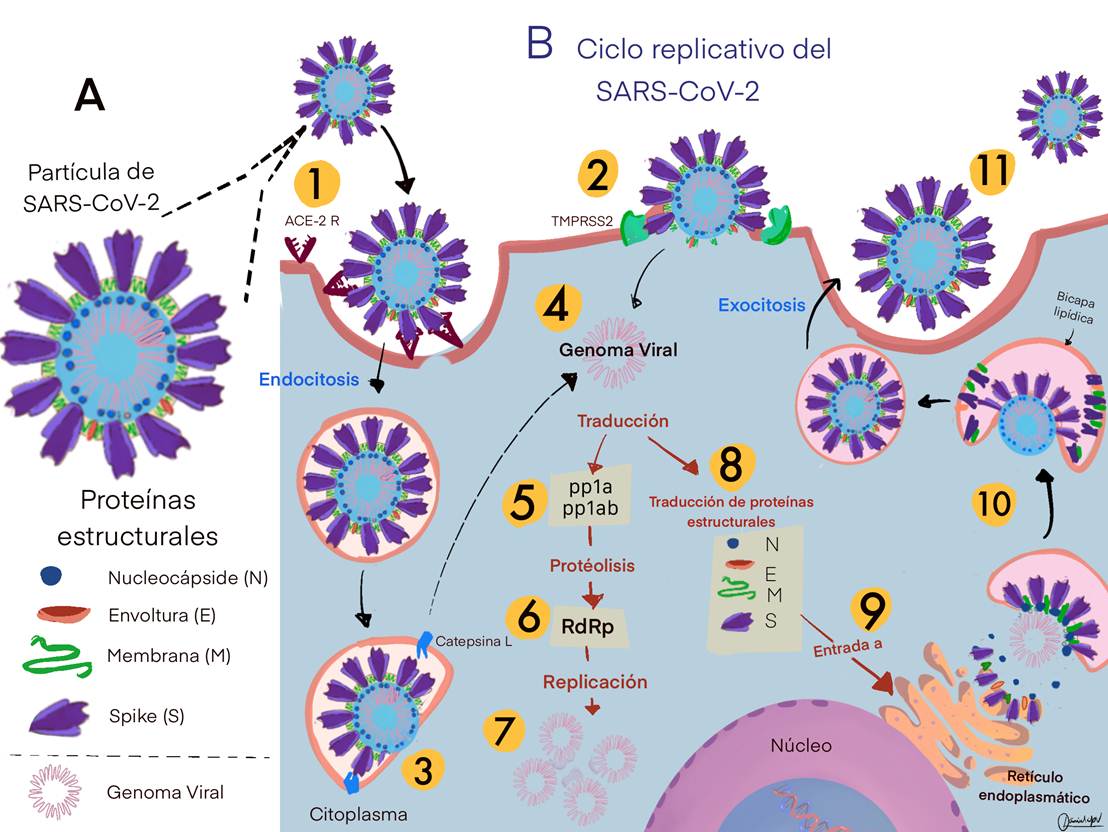
Figura 1. Estructura y ciclo replicativode SARS-CoV-2. A) Estructura del SARS-CoV-2 mostrando sus proteínas estructurales y el genoma en el interior. B) Ciclo replicativode SARS-CoV-2. 1) La proteína S (spike) se une al receptor ACE-2 en la superficie de los neumocitostipo II. 2) Rompimiento de la envoltura viral con la proteasa transmembranalde tipo serina2 o TMPRSS2. 3) Degradación de la envoltura con la proteasa catepsinaL. 4) Liberación del genoma viral. 5) Traducción de las poliproteínaspp1a y pp1ab, precursoras de la replicasaviral o ARN-polimerasa dependiente de ARN o RdRp, estas proteínas son procesadas por una (6) proteólisis para formar la replicasaviral (RdRp). 6) Ensamblaje de la RdRp. 7) Replicación del genoma viral. 8) traducción de proteínas estructurales. 9) Entrada de proteínas estructurales al retículo endoplásmico. 10) Ensamblaje del virus. 11) Exocitosisdel virus y muerte del neumocitotipo II. Figura modificada de: De VriesAAF,NethHeartJ. 2020 Jul;28(7-8):366-383.
2) Transcripción de la replicasa viral
Una vez que ha entrado el virus, se desbarata la cubierta de la cápside y se deja libre el genoma viral. Este genoma viral es una molécula monocatenaria positiva de ARN o [(+)g ARN], un ARN que puede ser traducida directamente en las proteinas virales deseadas. Así, el genoma de SARS-CoV-2 es traducido de inmediato en la célula hospedera. Alrededor de dos terceras partes del genoma consiste en dos marcos de lectura abiertos bastante extensos (ORF1a y ORF1b), los cuales son traducidos en dos poliproteinas llamadas pp1a y pp1ab (Fig.1). El tercio restante del genoma consiste en varios ORF que codifican para las proteinas estructurales (S, E, M, HE y N) (8). Las poliproteínas pp1a y pp1b van a ser cortadas por proteasas produciendo una serie de proteínas no estructurales o nsps (non-specific poly-proteins) las cuales en su conjunto forman el complejo viral replicasa. El complejo viral replicasa es un complejo muy importante para la replicación del virus. El complejo viral replicasa es una ARN polimerasa dependiente de ARN tambien conocida como RdRp (RNA-dependent RNA polymerase). Este complejo RdRp comienza a producir nuevas copias de genoma viral en sentido contrario tambien llamado antigenoma o (-)gARN(8).
El (-)gARN sirve como plantilla o molde para la producción de nuevos genomas virales por un proceso llamado replicación viral. El genoma (+)gARN, en cambio sirve como plantilla para la producción de las proteínas estructurales (S, M, E y N) y una serie de proteínas accesorias en el citoplasma de la célula. La RdRp es una enzima viral que tiene la propiedad de catalizar la replicación del ARN a partir de una plantilla de ARN, es decir sintetiza nuevas moléculas de ARN a partir de ARN. Esto contrasta con las típicas polimerasas dependientes de ADN, que todos los organismos celulares (incluyendo al humano) utilizan para catalizar la transcripción de ARN a partir de una platilla de ADN, que discutimos al inicio de este trabajo(8).
La RdRp es codificada por la región nsp12, mientras que la región nsp3 y nsp5 codifica para una proteasa similar a la papaina (PLPro) y para la proteasa principal (Mpro), las cuales rompen las poliproteinas pp1a y pp1ab para formar la RdRp. Varios medicamentos que actualmente se están fabricando van dirigidos a inhibir tanto a la RdRP como a estas proteasas(8,13).
3) Transcripción y replicación del genoma viral
Las múltiples copias del ARN del genoma viral de SARS-CoV-2 funcionan ahora como molde para que la replicasa viral (RdRp) empiece a sintetizar miles de copias del ARN viral(13).
4) Traducción de proteínas estructurales
Una vez que tenemos diversas copias de nuestro ARN viral, se comienza la traducción de las proteínas virales. Las proteínas virales estructurales transmembranales de la envoltura (S, HE, M y E) y otras proteinas accesorias son sintetizadas en el Retículo Endoplásmico Rugoso (RER), mientras que la proteína N de la nucleocápside es sintetizada por los ribosomas libres en el citoplasma. Algunas de estas proteínas tendrán algunas modificaciones posttraduccionales para obtener proteinas completamente maduras, principalemnte proteólisis(13).
5) Esamblaje y liberación del virión
Estudios elaborados con otros coronavirus indican que SARS-CoV-2 se ensambla en un compartimento intermedio entre el RER y el aparato de Golgi(13).La proteína N se une al (+)gARN recién formado. Las proteínas S, M, HE y E ingresan a esta estructura intermedia entre el RER y el aparato de Golgi. En este sitio las partículas virales se ensamblan en partículas completas, emergen y son transportadas en vesículas al exterior de la célula por exocitosis. Generalmente los virus no fabrican sus lípidos, en cambio utilizan los lípidos de la célula hospedera para su morfogénesis(13). Una vez liberados los nuevos virus se propagan para infectar a las células vecinas(15).
Ahora bien, cuando el neumocito es infectado, en un principio no reconoce ni al virus ni a su genoma como extraño, sin embargo posteriormente, empieza a sintetizar señales de auxilio antes de morir. Estas señales liberadas son principalmente mediadores de la respuesta inmune, que al ser liberadas son una señal para la infiltración de macrófagos y neutrófilos al área de infección (Fig.2). Los macrófagos a su vez, comienzan a producir interleucinas o IL (mensajeros químicos). Las IL-1, IL-6 y el factor de necrosis tumoral-alfa (TNF-µ) son las IL producidas por los macrófagos, las cuales cruzan la barrera epitelial y propician la vasodilatación del endotelio, incrementando la permeabilidad capilar(8,17). Conforme el daño va aumentando en las células alveolares, se va acumulando el líquido, lo que se conoce como edema intersticial(18)o edema alveolar(8). Este líquido hace más difícil el intercambio gaseoso, lo cual lleva a una hipoxia y a la dificultad para respirar (disnea)(8). (Fig.2)
Las citocinas sintetizadas tambien promueven la síntesis de procoagulantes en los capilares sanguíneos, promoviendo la formación de coágulos que llevan a su vez a una trombosis pulmonar y en casos más graves una trombosis sistémica(19). (Fig.2).
Por otra parte, los mediadores de la inflamación estimulan a los neutrófilos a liberar especies reactivas de Oxígeno (ROS) y diversas proteasas, las cuales en su conjunto dañan a todo el alveolo. Ante la baja producción de factor surfactante el alveolo colapsa(8). Las altas concentraciones de IL-1 e IL-6 viajan por el torrente sanguíneo hacia el sistema nervioso central, donde instruyen al hipotálamo para que libere prostaglandinas,las cuales causan un incremento en la fiebre corporal (Fig.2)(8).
Figura 2.
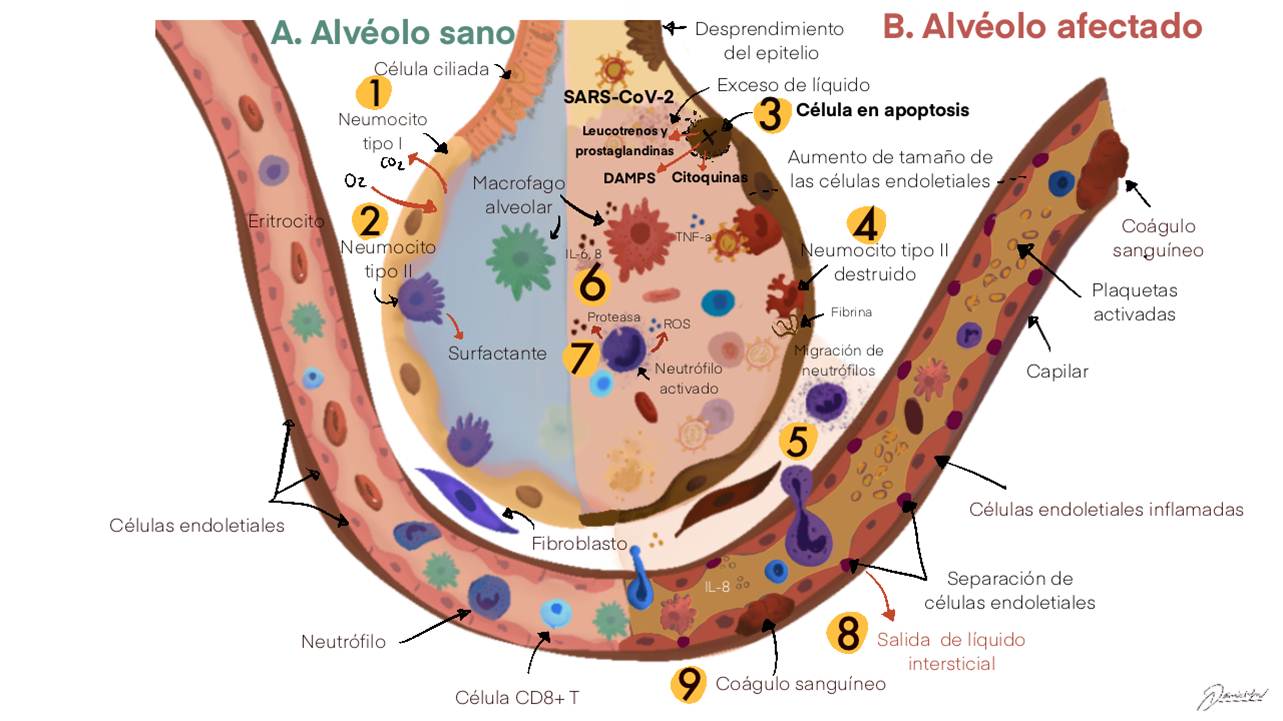
Figura 2. Fisiopatológiadel SARS-CoV-2. A) Del lado izquierdo de la figura se representa un alveolo sano con sus dos tipos de células alveolares: los neumocitostipo I y tipo II. Los neumocitostipo I (1) forman un epitelio plano simple encargado del intercambio gaseoso, mientras que los tipo II (2) son células cúbicas cuya función es la secreción de factor surfactante. B) Representación esquemática del daño a nivel alveolar por la infección con SARS-CoV-2. Durante la infección por SARS-CoV-2 en el alveolo, los neumocitotipo II antes de morir secretan una serie de señales de auxilio (4) incluyendo leucotrienosque propician la inflamación, las prostaglandinas, las cuales salen al torrente circulatorio y estimulan al sistema nervioso central para aumentar la temperatura corporal y patrones moleculares asociados al daño o DAMPS o señales de peligro. Todas las señales de auxilio atraviesan la barrera hemato-gaseosa para llegar a los vasos sanguíneos y funcionan como quimioatrayentesde neutrófilos (5). A su vez los macrófagos alveolares producen a su vez una serie de citocinaspara atraer a los neutrófilos al sitio (6). Posteriormente la célula muere (4) causando una mayor inflamación. Los neutrófilos a su vez producen proteasas y especies reactivas de Oxígeno o ROS (7), los cuales destruyen a todo el tejido dañado, tanto a las células sanas como las dañadas. Las citocinasen el vaso sanguíneo estimula la separación del endotelio, filtrando líquido intersticial, provocando un edema intersticial y posteriormente un edema alveolar (8). Las citocinastambienaumentan la síntesis de procoagulantesen la sangre, aumentando la coagulación y la formación de coágulos y promoviendo una trombosis pulmonar. Figura modificada de: MachhiJ, et al, J NeuroimmunePharmacol. 2020 Sep;15(3):359-386.
Conforme incrementa la permeabilidad capilar el volúmen sanguíneo disminuye, lo que lleva a una hipotensión y una disminución en la perfusión de diversos órganos, lo que se conoce como fallo multiorgánico. Cuando hay fallo sistémico se elevan las concentraciones sanguíneas de urea, nitrógeno y creatinina en el riñon. El hígado tambien libera mediadores inflamatorios y biomoléculas tales como: aspartato transaminasa, alanina transaminasa, bilirrubina, proteína C reactiva, fibrinógeno e IL-6, las cuales pueden ser utilizadas como biomarcadores diagnósticos de COVID-19 (8).Este cuadro puede derivar entonces en insuficiencia respiratoria que requiere ventilación mecánica y si no es tratado puede llevar a la muerte. En la Figura 2 se resumen los eventos fisiopatológicos del COVID-19(13).
Pruebas diagnósticas.
Actualmente hay una demanda para que los métodos diagnósticos de COVID-19 sean más sensibles y específicos. Las pruebas diagnósticas utilizadas por el momento para ver la presencia del virus se pueden dividir en cuatro tipos: 1) cultivo de SARS-CoV-2, 2) detección de genoma viral, 3) detección de antígenos virales y; 4) detección de anticuerpos. Otras técnicas no específicas como las radiografías para visualizar el daño pulmonar, análisis de biomarcadores para verificar el daño de tejidos y órganos e inflamación, microscopía electrónica o ensayos infecciosos virales, también pueden ayudar en el diagnóstico de COVID-19. Cada una de estas pruebas tiene sus ventajas y desventajas.
Para el cultivo de virus, se requieren laboratorios de bioseguridad clase 3 con una infraestructura costosa, lo cual no es muy común en la mayoría de los laboratorios de análisis clínicos y microbiológicos. Por lo que el cultivo de SARS-CoV-2 generalmente se realiza en laboratorios de investigación(20). Los estuches o kits comerciales para la detección de antígenos, requieren a su vez de la preparación de anticuerpos monoclonales y policlonales, lo cual implica un proceso complicado que requiere de mucho tiempo.
La detección del genoma viral utilizando la reacción en cadena de la polimerasa en tiempo real es la técnica más utilizada para diagnosticar COVID-19. A continuación se detalla el proceso diagnóstico utilizado por la Secretaría de Salud en México(20).
Prueba para la detección del genoma de SARS-CoV-2.
La prueba de laboratorio más utilizada para el diagnóstico de COVID-19 es la reacción en cadena de la polimerasa en tiempo real cuantitativa (RT-qPCR). La RT-qPCR es un procedimiento de varios pasos que permite la detección del material genético en las vías respiratorias altas y bajas. Se basa principalmente en cuantificar la presencia de genes virales tales como el gen de la RdRp, de la secuencia de ORF1ab, del gen de la proteína E, N o S del genoma de SARS-CoV-2(21). Se resume en los siguientes pasos:
1) Toma de la muestra. La muestra deberá tomarse de manera obligatoria por personal designado por la unidad de salud y capacitado en el uso de equipo de protección personal. Las muestras deberán ser consideradas como altamente infecciosas(20).
Se toman dos muestras mediante hisopado, introduciendo un hisopo largo de rayón, dacrón o nylon por la boca o nariz para hacer un pequeño raspado en la orofaringe y otro en la nasofaringe. Ambos hisopos se sumergen en un tubo hermético con medio de transporte viral. Se almacena a 4oC y es transportado al laboratorio. Cabe señalar que el éxito del diagnóstico virológico depende principalmente de la calidad de la muestra, así como de las condiciones de su transporte y almacenamiento antes de ser procesada en el laboratorio(20).
2) Acondicionamiento de la muestra. Una vez recibida la muestra en el laboratorio se verifica que cumpla con los requisitos mínimos de calidad y que cumpla con la definición operacional de un caso sospechoso emitido por la Secretaría de Salud(20).
3) Extracción y purificación del genoma viral. Esto incluye: lisis del virus y lavados del ARN. En el primer paso,se desintegra la envoltura y la cápside viral con detergentes y proteasas grado molecular, proceso conocido como lisis (rompimiento), dejando expuesto el genoma viral. Este paso es importante debido a la presencia ubicua de enzimas ribonucleasas en las células y tejidos, las cuales pueden degradar rápidamente el ARN. Para la purificación, se agrega un alcohol (etanol o isopropanol) a la mezcla y se hace pasar por una columna, donde el ARN quedará unido a una membrana sílica de la columna. Posteriormente se hacen varios lavados y por último, se agrega un amortiguador de elución para remover el ARN de la membrana y para ser colectado en un tubo contenedor, donde se almacenará a -20oC hasta su uso posterior(20).
4) Transcripción Reversa. Debido a que el genoma viral es un ARN, el siguiente paso es retrotranscribir la hebra de ARN en una molécula de ADN complementario, usando una enzima llamada transcriptasa reversa. Esta enzima, obtenida de un retrovirus, tiene como función sintetizar ADN utilizando como plantilla una molécula de ARN monocatenario. Su nombre obedece a que el proceso normal de la transcripción, sucede cuando se sintetiza una molécula de ARN a partir de una secuencia inicial de ADN y no al revés.
5) Reacción en cadena de la polimerasa. En este paso se elaboran varias copias de genes específicos del SARS-CoV-2 en el ADN obtenido mediante la retrotranscripción. De esta manera amplificamos la señal. Estas copias se realizan repitiendo varias veces (hasta 40 ciclos) la replicación del ADN con una enzima termoestable llamada ADN polimerasa. Mediante un método basado en fluorescencia se podrá observar la presencia o ausencia del material genético. Para profundizar en puede consultar el siguiente video(21): https://www.youtube.com/watch?v=ThG_02miq-4
6) Análisis de resultados: Cuando la señal de una muestra de un paciente con sospecha de COVID-19 se amplifica de manera logarítmica decimos que el resultado es positivo. Si la señal no amplifica decimos que el resultado es negativo.
Las pruebas de PCR tienen la ventaja de ser altamente específicas y con una gran sensibilidad, sin embargo, requieren de personal altamente especializado para minimizar cualquier contaminación, así como se requiere de infraestructura costosa. A su vez los tiempos de resultados son relativamente largos, siendo un factor limitante cuando se deben procesar un gran número de muestras.
Algunos estudios demuestran que la sensibilidad del ensayo puede rondar entre 30 y 60% dependiendo del tipo de muestra, la condición del paciente y el protocolo empleado(23). Por lo que, para incrementar la sensibilidad es recomendable utilizar diversas pruebas(23).
Actualmente se encuentran en desarrollo dispositivos biosensores, constituídos por un microchip sensor en contacto con moléculas biológicas selectivas, (muy parecidos a los sensores de glucosa). La captura de la molécula diana produce cambios fisicoquímicos que son detectados por el transductor y procesados en un valor numérico cuantificable. Para la detección genómica es posible usar biosensores ópticos para la detección directa del ARN viral, sin embargo, esta tecnología no se encuentra actualmente en el mercado(24).
Pruebas de detección de antígenos virales
Otro método para confirmar una infección activa del SARS-CoV-2 es el ensayo por inmunoadsorción ligado a enzimas o ELISA (Enzyme-Linked Immunosorbent Assay). Este ensayo detecta ciertas proteínas virales (antígenos) en muestras de pacientes. Se usa un hisopo para tomar una muestra del fluido de la nariz o la garganta y se conserva en un medio de transporte viral. Posteriormente en el laboratorio se lleva a cabo la lisis del virus. La mezcla se deposita en un pocillo previamente recubierto con un anticuerpo específico para las proteínas virales. Si el anticuerpo reconoce algún epítope del antígeno de la muestra, se unirá a él. Después de un lavado se aplica un segundo anticuerpo conjugado con una enzima colorimétrica. Un sustrato incoloro es agregado posteriormente, el cual será convertido en un producto colorido que podrá ser detectado por un colorímetro. La cantidad de color formado será proporcional con la cantidad de virus presente en la muestra. Debido a que las pruebas de ELISA son procedimientos laboriosos que requieren anticuerpos altamente específicos y costosos no son utilizados(25).
Pruebas de detección de anticuerpos
Este tipo de pruebas detectan la presencia de anticuerpos en al plasma sanguíneo, lo cual indica indirectamente la respuesta inmune adaptativa. Ante una infección, el sistema inmune de una persona responde desencadenando la producción de anticuerpos para atacar la infección y confiriendo cierta inmunidad ante posteriores reinfecciones. Los anticuerpos generados por el organismo suelen tener como diana, determinantes antigénicos clave en el agente patógeno, por ejemplo, las proteínas estructurales del virus(26).
En estas pruebas, se pone en contacto la sangre del paciente con los antígenos del virus de manera que la presencia de anticuerpos en el suero es detectada. Se realizan con tiras inmunocromatográficas coloidales marcadas con oro, similares a las pruebas comerciales de embarazo. Hay dos tipos de pruebas, una que detecta la cantidad de anticuerpos totales y otra que detecta la presencia de inmunoglobulinas M (IgMs) e inmunoglobulinas G (IgGs) anti-SARS-CoV-2. Las primeras no diferencían IgMs o IgGs, por lo que los resultados no determinan la evolución de la enfermedad. Las segundas funcionan tambien con tiras de nitrocelulosa pero en este caso contienen tres líneas, dos para la detección de anticuerpos, (una línea para la detección de IgMs y otra para IgGs) y una tercera línea para el control de calidad. En la línea IgM se han fijado previamente anticuerpos monoclonales anti-IgM humanos anti-SARS-CoV-2, mientras que la línea IgG se fija un reactivo para detectar los IgG anti-SARS-CoV-2(27,28). Una ventaja es que se puede diferenciar las distintas etapas de la infección por los distintos tipos de anticuerpos que se producen: por ejemplo, las IgM se generan al principio, y representan un proceso de infección aguda, mientras que, las IgG, indican una infección primaria o una respuesta a la fase aguda de infecciones secundarias(26). Para profundizar consulte el siguiente video(29): https://www.youtube.com/watch?v=ouDY_pxGiIY
Las pruebas serológicas tienen la ventaja de ser rápidas y de bajo costo, debido a que requieren muy poca muestra son mínimamente invasivas, pueden ser realizadas en el sitio de toma de muestra y no requiere de instrumentación compleja o de personal altamente especializado. Sin embargo, presentan una sensibilidad muy limitada con una alta posibilidad de falsos negativos y tiene una variabilidad inherente a la respuesta inmune de cada individuo. La sensibilidad de estas pruebas varía dependiendo de la casa comercial(27,28).
CONCLUSIONES
El brote de SARS-CoV-2 ha sido una amenaza mundial. La información respecto al virus sigue siendo limitada e inconsistente, aunado a que cada día sale nueva información. Los investigadores se esfuerza para sacar nueva información día con día para producir una tratamiento o una vacuna. En este trabajo presentamos una breve revisión de la fisiopatología del SARS-CoV-2 y su diagnóstico actual. Aun se requiere esfuerzo por parte del sector salud para esclarecer de manera más puntual la fisiopatología del virus para poderlo implementar en mejores técnicas diagnósticas y tratamientos. Pero, mientras no tengamos una vacuna o un tratamiento adecuado es importante continuar las medidas de higiene como el uso de tapabocas, la distancia social, el lavado de manos y el seguimiento a los lineamientos establecidos por la Secretaría de Salud. Esto nos ayudará a reducir la propagación del virus.
REFERENCIAS
- Alberts B, Johnson A, Lewis J, Raff M, Roberts K y Walter P. Molecular Biology of the Cell. 4th edition. New York: Garland Science; 2002. Genetic Information in Eucaryotes. Available from: https://www.ncbi.nlm.nih.gov/books/NBK26909/
- Alberts B, Johnson A, Lewis J, Raff M, Roberts K y Walter P. Molecular Biology of the Cell. 4th edition. New York: Garland Science; 2002. How Genomes Evolve. Available from: https://www.ncbi.nlm.nih.gov/books/NBK26836/
- HardisonRC.Comparative Genomics. PLoS Biol. 2003;1(2): e58. https://doi.org/10.1371/journal.pbio.0000058
- Alberts B, Johnson A, Lewis J, Raff M, Roberts K y Walter P. Molecular Biology of the Cell. 4th edition. New York: Garland Science; 2002. Chapter 6, How Cells Read the Genome: From DNA to Protein. Available from:https://www.ncbi.nlm.nih.gov/books/NBK21050/
- Uzawa T, Yamagishi A, Oshima T. Polypeptide synthesis directed by DNA as a messenger in cell-free polypeptide synthesis by extreme thermophiles, Thermus thermophilus HB27 and Sulfolobus tokodaii strain 7. J Biochem. 2002 Jun;131(6):849-53. doi: 10.1093/oxfordjournals.jbchem.a003174.
- Alberts B, Johnson A, Lewis J, Raff M, Roberts K y Walter P. Molecular Biology of the Cell. 4th edition. New York: Garland Science; 2002. Introduction to Pathogens. Available from: https://www.ncbi.nlm.nih.gov/books/NBK26917/
- Wu F, Zhao S, Yu B, Chen YM, Wang W, Song ZG, et al. A new coronavirus associated with human respiratory disease in China. Nature. 2020 Mar;579(7798):265- doi: 10.1038/s41586-020-2008-3. Epub 2020 Feb 3. Erratum in: Nature. 2020 Apr;580(7803):E7.doi: 10.1038/s41586-020-2202-3.
- Fadaka AO, Sibuyi NRS, Adewale OB, Bakare OO, Akanbi MO, Klein A. Understanding the epidemiology, pathophysiology, diagnosis and management of SARS-CoV-2. J Int Med Res. 2020 Aug;48(8):300060520949077. doi: 10.1177/0300060520949077.
- Organización Mundial de la Salud. Zoonosis y medio ambiente. Fecha de acceso: 19 de Octubre del 2020. Disponible en: https://www.who.int/foodsafety/areas_work/zoonose/es/
- Kang S, Yang M, Hong Z, Zhang L, Huang Z, Chen X, et al. Crystal structure of SARS-CoV-2 nucleocapsid protein RNA binding domain reveals potential unique drug targeting sites. Acta Pharm Sin B. 2020 Jul;10(7):1228-1238. doi: 10.1016/j.apsb.2020.04.009.
- Zalzala HH. Diagnosis of COVID-19: facts and challenges. New Microbes New Infect. 2020 Nov;38:100761. doi: 10.1016/j.nmni.2020.100761.
- Perrotta F, Matera MG, Cazzola M, Bianco A. Severe respiratory SARS-CoV2 infection: Does ACE2 receptor matter? Respir Med. 2020 Jul;168:105996. doi: 10.1016/j.rmed.2020.105996.
- Machhi J, Herskovitz J, Senan AM, Dutta D, Nath B, Oleynikov MD, et al. The Natural History, Pathobiology, and Clinical Manifestations of SARS-CoV-2 Infections. J Neuroimmune Pharmacol. 2020 Sep;15(3):359-386. doi: 10.1007/s11481-020-09944-5.
- Ortiz ME, Thurman A, Pezzulo AA, LeidingerMR, Klesney-TaitJA, KarpPH,et al. Heterogeneous expression of the SARS-Coronavirus-2 receptor ACE2 in the human respiratory tract. EBioMedicine. 2020;60:102976. Published 2020 Sep 21. doi:10.1016/j.ebiom.2020.102976
- de Vries AAF. SARS-CoV-2/COVID-19: a primer for cardiologists. Neth Heart J. 2020 Jul;28(7-8):366-383. doi: 10.1007/s12471-020-01475-1.
- Meyerowitz EA, Richterman A, Gandhi RT, Sax PE. Transmission of SARS-CoV-2: A Review of Viral, Host, and Environmental Factors. Ann Intern Med. 2020;M20-5008. Published 2020 Sep 17. doi:10.7326/M20-5008
- Libby P, Lüscher T. COVID-19 is, in the end, an endothelial disease. Eur Heart J. 2020 Sep 1;41(32):3038-3044. doi: 10.1093/eurheartj/ehaa623.
- Zhao Y, Zhao Z, Wang Y, Zhou Y, Ma Y, Zuo W. Single-Cell RNA Expression Profiling of ACE2, the Receptor of SARS-CoV-2. Am J Respir Crit Care Med. 2020 Sep 1;202(5):756-759. doi: 10.1164/rccm.202001-0179LE.
- Divani AA, Andalib S, Di Napoli M, Lattanzi S, Hussain MS, Biller J, et al. Coronavirus Disease 2019 and Stroke: Clinical Manifestations and Pathophysiological Insights. J Stroke Cerebrovasc Dis. 2020 Aug;29(8):104941. doi: 10.1016/j.jstrokecerebrovasdis.2020.104941.
- Secretaría de Salud. Lineamiento estandarizado para la vigilancia epidemiológica y por laboratorio de enfermedad respiratoria viral. México: Secretaría de Salud; Agosto, 2020.
- Corman VM, Eckerle I, Bleicker T, Zaki A, Landt O, Eschbach-Bludau M, et al. Detection of a novel human coronavirus by real-time reverse-transcription polymerase chain reaction. Euro Surveill. 2012 Sep 27;17(39):20285. doi: 10.2807/ese.17.39.20285-en. Erratum in: Euro Surveill. 2012;17(40): pii/20288.
- Biology with animations: Coronavirus test: Real Time RT-PCR animation video.2020, fecha de acceso: 19 de Octubre 2020. Disponible en: https://www.youtube.com/watch?v=ThG_02miq-4
- Meng QB, Peng JJ, Wei X, Yang JY, Li PC, Qu ZW, et al. Clinical application of combined detection of SARS-CoV-2-specific antibody and nucleic acid. World J Clin Cases. 2020 Oct 6;8(19):4360-4369. doi: 10.12998/wjcc.v8.i19.4360.
- Shabani E, Dowlatshahi S, Abdekhodaie MJ. Laboratory detection methods for the human coronaviruses. Eur J Clin Microbiol Infect Dis. 2020 Sep 28:1–22. doi: 10.1007/s10096-020-04001-8.
- Li Guo, Lili Ren, Siyuan Yang, Meng Xiao, De Chang, Fan Yang, et al. Profiling Early Humoral Response to Diagnose Novel Coronavirus Disease (COVID-19), ClinInfectDis.2020 1 august; 71(15):778–85, https://doi.org/10.1093/cid/ciaa310
- Deeks JJ, Dinnes J, Takwoingi Y, Davenport C, Spijker R, Taylor-Phillips S, et al. Cochrane COVID-19 Diagnostic Test Accuracy Group. Antibody tests for identification of current and past infection with SARS-CoV-2. Cochrane Database Syst Rev. 2020 Jun 25;6(6):CD013652. doi: 10.1002/14651858.CD013652.
- Zainol Rashid Z, Othman SN, Abdul Samat MN, Ali UK, Wong KK. Diagnostic performance of COVID-19 serology assays. Malays J Pathol. 2020 Apr;42(1):13-21.
- Zalzala HH. Diagnosis of COVID-19: facts and challenges. New Microbes New Infect. 2020;38:100761. doi:10.1016/j.nmni.2020.100761
- Biology with animations: Coronavirus antibody tests animation video.Año de publicación: 2020, fecha de acceso: 19 de Octubre 2020. Disponible en: https://www.youtube.com/watch?v=ouDY_pxGiIY





